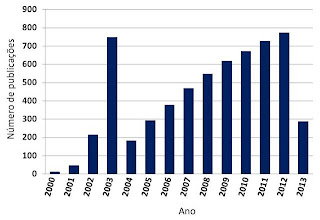
Figura 1: Evolução do número de publicações científicas indexadas e
acessíveis no portal do NCBI, de 2000 a 2013. Observa-se uma clara tendência a
aumento sustentado, indicando o interesse que este assunto desperta na
comunidade científica. A busca foi feita com a palavra chave siRNA. A adição de
outras palavras chave relacionadas ao assunto (dsRNA, RNA dependent RNA polymerase, etc) eleva o número total de publicações para mais de 50 mil.
Em algumas postagens deste portal e em outros documentos
técnicos explicamos como os pequenos RNA são gerados e como interferem na
expressão de um gene (http://genpeace.blogspot.com.br/2011/09/entre-seriedade-e-o-disparate.html; http://www.ufpe.br/biolmol/iRNA.htm) e de que forma a avaliação de risco de produtos baseados nesta
tecnologia é feita nestes casos (http://cienciahoje.uol.com.br/blogues/bussola/2013/04/dsRNA%20-Flavio%20Finardi.pdf; http://genpeace.blogspot.com.br/2013/04/rota-ao-dano-pelo-rna-de-interferencia.html).
Entretanto, tem-se lido frequentemente em portais que se
posicionam contra a biotecnologia agrícola a afirmação de que pequenos RNA de
interferência inesperados (“unexpected” ou “unanticipated”) podem ser criados em plantas transgênicas, dirigidos contra
alvos não intencionais. Nestas postagens isso é passado como um risco sério, que
não foi avaliado pelas agências de governo do Mundo responsáveis pela segurança
dos OGM. Não nos parece, contudo, que a probabilidade seja alta ou que acarrete
algum problema ambiental ou de saúde, particularmente no caso do feijão Embrapa
5.1, e assim vamos contestar a hipótese; entretanto, nunca se pode saber
exatamente do que os opositores intransigentes da biotecnologia estão falando,
porque raramente são claros em suas explicações, sempre baseadas em hipóteses
muito imaginativas. Acreditamos que estejam falando do “PCR degradativo”
(Liparti etal., 2001), em que um siRNA pode atuar como primer para a síntese da segunda fita do RNA por uma RNA polimerase
dependente de RNA, encontrada em plantas.
Antes de adentrar neste assunto, gostaríamos de relembrar a
construção genética do feijão GM da Embrapa, detalhadamente descrita nas várias
publicações do grupo de pesquisa responsável pelo seu desenvolvimento (Aragão
et al. 2013 e referências incluídas nele). A ideia que dirigiu os estudos foi a
inibição da produção da enzima Rep (replicase) pelo mecanismo de interferência
de RNA. Para tal o feijoeiro foi transformado com uma construção contendo um
segmento de DNA no qual duas cópias invertidas de 411 pb aparecem separadas por
um trecho não repetido de 750 pb. A transcrição deste trecho, a partir de um
promotor forte, produz grampos de RNA fita dupla no núcleo, que são
transportados para o citoplasma via exportina e clivados em dsRNA pela enzima
Dicer. Estes fragmentos irão direcionar o complexo enzimático Argonauta para o
mRNA específico que codifica a replicase (Figura 2).
Figura 2: (A) Grampo
de RNA produzido no núcleo das células do feijoeiro a partir da transcrição do
cassete de expressão introduzido na variedade transgênica Embrapa 5.1. Uma vez
produzido, o grampo é exportado por um transportador específico para o
citoplasma, onde será clivado pelas enzimas Dicer. (B)
A “cabeça” ou “loop” é separada do
pescoço, que é subsequentemente clivado em pequenos pedaços de RNA de fita
dupla, que depois são convertidos em pequenos RNA de fita simples. Os trechos
de RNA mais longos são degradados e uma fita dos pequenos RNA é carregada em um
complexo proteico contendo a enzima Argonauta, que cliva os mRNA que têm
complementaridade (neste caso, os genes para AC1). Sem o produto destes
mensageiros o vírus não pode se multiplicar e a planta se torna resistente ao
vírus.
Entretanto, o processo de degradação do mRNA não está restrito a
uma única quebra pela enzima Argonauta: graças à ação de uma RNA polimerase
dependente de RNA e do pareamento de pequenos RNA fita simples derivados dos
dsRNA gerados pela Dicer, o restante do mRNA acima do ponto de corte pode ser
transformado em fita dupla, gerando consequentemente um novo alvo para a Dicer,
que o corta em pequenos fragmentos. Estes pequenos fragmentos são o resultado
final da degradação do mRNA, mas também podem ser carregados no Argonauta para
um outro round de clivagens simples
de mRNA.
O processo todo está representado na Figura 3 a seguir. Ela
mostra o mecanismo real pelo qual um siRNA gera a degradação de toda a região a
montante de seu alvo no mRNA específico. Inicialmente é preciso saber que o
mRNA transcrito a partir do genoma viral é policistrônico, ou subgenômico, como
algumas vezes é chamado, contendo os transcritos dos genes AC1, AC2 e AC3, que
codificam para as proteínas Rep, TrAP e REn (http://viralzone.expasy.org/all_by_species/111.html) (Figura 3A). Já sabemos também que a construção inserida no genoma do
feijão tem um par de trechos idênticos de 411 pares de base do gene AC1,
dispostos em sentidos opostos e separados entre si por um trecho não repetido
de pouco mais de 700 pb, tomado de um íntron de planta (Figura 3B). Por isso, o
alvo primário do complexo Argonauta será a região que tem o transcrito do gene
AC1, que fica mais à montante do mRNA. Os demais genes estão abaixo dele, no
mRNA. Como já comentamos, o processo começa quando o dsRNA gerado no núcleo,
através da transcrição da construção inserida no organismo, é transportado para
o citoplasma e quebrado em pequenos RNA fita dupla pela enzima Dicer (Figura
3C). Uma fita simples de cada pequeno RNA, carregada no complexo enzimático
Argonauta, serve de “sonda” para encontrar o mRNA específico para o qual a
construção foi dirigida. Neste caso, os mRNAs policitrônicos serão cortados em
algum ponto dentro da sequência equivalente a AC1, representada em verde
bandeira na figura 1A.
Figura 3: Esquema representativo dos genes AC1, AC2 e AC3 de BGMV,
seu transcrito, e os vários passos que levam à degradação completa do mRNA
acima do alvo definido pela construção genética.
O segundo passo na degradação do mRNA (ao menos da região acima,
isto é, 5´ do corte dado pelo Argonauta) vai depender do pareamento de uma pequena
fita simples, gerada a partir dos dsRNA, com o mRNA policistrônico. Numa
primeira etapa, esta fita simples só pode parear nas regiões de
complementaridade com a construção (indicadas em verde bandeira na Figura 3D),
isto é, a montante do mRNA. A partir deste primer
uma RNA polimerase RNA dependente pode estender a fita simples até a extremidade
5´ do mRNA, formando um trecho fita dupla. Este, por sua vez, servirá de
substrato para Dicer e gerará finalmente novos siRNA diferentes daqueles que
são gerados pelo processamento direto do dsRNA transgênico. Obviamente isso não
tem nada de “unintended” e fica restrito às sequencia do RNA alvo. E
mais ainda: fica restrito à região a montante (5´) do alvo, portanto , dentro
do gene AC1 e sua região 5´ não traduzida. Não há qualquer possibilidade de
geração de siRNA com similaridade a outros genes se apenas este mRNA estiver
disponível.
Para que os demais genes possam gerar siRNA é necessários que
estejam acima (isto é, 5´) do gene AC1. Isso só poderia acontecer caso um longo
transcrito fosse produzido, no sentido inverso,a partir do promotor do gene
para CP (veja figura 4, a seguir). Não é isso que deve acontecer na transcrição
deste DNA, pois os genes abaixo do CP estão em sentido invertido e não poderiam
ser expressos, fazendo de tal transcrito um “peso metabólico” desnecessário e
perigoso para a sobrevivência do vírus. Um transcrito tão longo nunca foi
descrito na literatura especializada, nem para o BGMV nem para vírus
semelhantes.
Figura 4: Genoma duplo do vírus do mosaico dourado do feijoeiro
(BGMV) (Fonte: Swiss Institute of Bioinformatics). O DNA-A dá origem a dois
transcritos: no sentido anti-horário o mRNA policistrônico tem o transcrito dos
genes AC1, AC2 e AC3 (codificando para as proteínas Rep, TrAP, e REn) e no
sentido antihorário o mRNA tem o gene para CP.
Em resumo, a construção inserida no feijão Embrapa 5.1 não
parece permitir a geração de siRNA dirigidos a outro gene que não o AC1 e sua
região 5´ no genoma do vírus. Todo o resto é especulação. Ainda assim, a equipe
da Embrapa procurou e não encontrou evidências de outros siRNA, como mencionado
nas publicações.
Podemos concluir que é fantasiosa a ideia de que siRNA
inesperados sejam produzidos neste feijão e, mais ainda, que tenham qualquer importância
no aumento de um pretenso risco à saúde humana ou animal, uma vez que a rota ao
dano para este caso é de uma probabilidade de ocorrência insignificante e que
os possíveis danos são ainda imponderáveis (veja-se a postagem http://genpeace.blogspot.com.br/2013/04/rota-ao-dano-pelo-rna-de-interferencia.html
mencionada anteriormente).
Referências
Aragão FJ, Nogueira EO, Tinoco ML, Faria JC. (2013) - Molecular characterization of the
first commercial transgenic common bean immune to the Bean golden mosaic virus.
J Biotechnol. doi: 10.1016/j.jbiotec.2013.04.009. [Epub ahead of print]
Lipardi C, Wei Q, Paterson BM (2001)- RNAi as random degradative PCR:
siRNA primers convert mRNA into dsRNAs that are degraded to generate new
siRNAs. Cell 107(3):297-307.



Nenhum comentário:
Postar um comentário